Genómica tumoral: construcción histórica y perspectivas para el futuro
Tumor genomics: historical development and perspectives for the future
Cómo citar
Descargar cita

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
Mostrar biografía de los autores
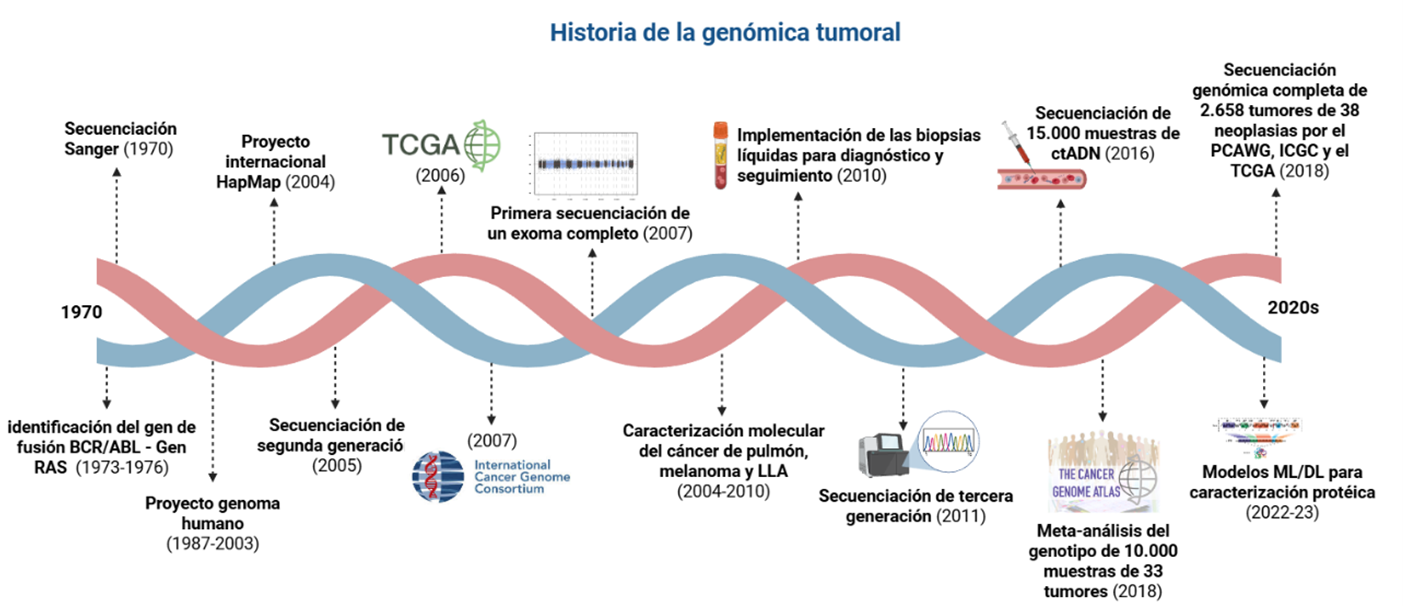
Introducción: desde el descubrimiento del cromosoma Filadelfia hasta la llegada de las secuenciaciones de nueva generación (NGS), la genómica oncológica ha transformado la comprensión molecular del cáncer. Este artículo presenta una revisión narrativa con reconstrucción histórica, hitos fundamentales, principales barreras y perspectivas futuras en genómica tumoral.
Métodos: se realizó una búsqueda sistematizada en tres bases de datos internacionales. De 137 registros, se seleccionaron 29 artículos para revisión completa, además de diversas fuentes históricas primarias.
Resultados: este trabajo examina la evolución tecnológica, los hitos clínicos y el impacto de la genómica en la práctica oncológica. Además, se mencionan algunos desafíos actuales y perspectivas para su implementación en el futuro.
Discusión: a pesar de revisar un rango temporal de poco más de medio siglo, el equipo de investigación reconoce que estos conceptos aún están en desarrollo y que, superar las barreras genómicas actuales probablemente será el hito para considerar en el futuro. La oncogenómica podría estar en una era dorada, su implementación clínica es cada vez más una realidad y un estándar.
Conclusión: en la era del análisis masivo de datos, las inteligencias artificiales y los consorcios robustos de colaboración global, mirar críticamente al pasado también puede fortalecer los pasos futuros. El cáncer ya se comprende como una enfermedad del genoma; no obstante, es necesario seguir superando obstáculos teóricos, científicos, de implementación, accesibilidad y transversalidad.
Visitas del artículo 0 | Visitas PDF 0
Descargas
- Stafford N. Janet Rowley. BMJ [Internet]. 2014;348. Disponible en: https://doi.org/10.1136/bmj.g447
- Fonseca-Montaño MA, Blancas S, Herrera-Montalvo LA, Hidalgo-Miranda A. Cancer Genomics. Arch Med Res. [Internet]. 2022;53(8):723-31. Disponible en: https://doi.org/10.1016/j.arcmed.2022.11.011
- Yates LR, Campbell PJ. Evolution of the cancer genome. Nat Rev Genet. [Internet]. 2012;13(11):795-806. Disponible en: https://doi.org/10.1038/nrg3317
- Lee-Six H. Somatic evolution in human blood and colon. Doctoral tesis University of Cambridge. 2018. Disponible en: https://doi.org/10.17863/CAM.37059
- Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature. [Internet]. 2009;458(7239):719-24. Disponible en: https://doi.org/10.1038/nature07943
- Nowell C. The minute chromosome (Ph1) in chronic granulocytic leukemia. Blut. [Internet]. 1962;8(2):65-6. Disponible en: https://doi.org/10.1007/bf01630378
- Rowley JD. A New Consistent Chromosomal Abnormality in Chronic Myelogenous Leukaemia identified by Quinacrine Fluorescence and Giemsa Staining. Nature. [Internet]. 1973;243(5405):290-3. Disponible en: https://doi.org/10.1038/243290a0
- Groffen J, Stephenson J, Heisterkamp N, Deklein A, Bartram C, Grosveld G. Philadelphia chromosomal breakpoints are clustered within a limited region, bcr, on chromosome 22. Cell. [Internet]. 1984;36(1):93-9. Disponible en: https://doi.org/10.1016/0092-8674(84)90077-1
- Spector DH, Smith K, Padgett T, McCombe P, Roulland-Dussoix D, Moscovici C, et al. Uninfected avian cells contain RNA related to the transforming gene of avian sarcoma viruses. Cell. [Internet]. 1978;13(2):371-9. Disponible en: https://doi.org/10.1016/0092-8674(78)90205-2
- Shampo MA, Kyle RA. J. Michael Bishop—Nobel Laureate in Medicine or Physiology. Mayo Clinic Proceedings. [Internet]. 2002;77(12):1312. Disponible en: https://doi.org/10.4065/77.12.1312
- Nowell PC. The Clonal Evolution of Tumor Cell Populations: Acquired genetic lability permits stepwise selection of variant sublines and underlies tumor progression. Science. [Internet]. 1976;194(4260):23-8. Disponible en: https://doi.org/10.1126/science.959840
- Rowley JD. Chromosome abnormalities in leukemia. J Clin Oncol. [Internet]. 1988;6(2):194-202. Disponible en: https://doi.org/10.1200/JCO.1988.6.2.194
- Ribeiro IP, Melo JB, Carreira IM. Cytogenetics and Cytogenomics Evaluation in Cancer. IJMS. [Internet]. 2019;20(19):4711. Disponible en: https://doi.org/10.3390/ijms20194711
- Mullis K, Faloona F, Scharf S, Saiki R, Horn G, Erlich H. Specific Enzymatic Amplification of DNA In Vitro: The Polymerase Chain Reaction. Cold Spring Harbor Symposia on Quantitative Biology. [Internet]. 1986;51(0):263-73. Disponible en: https://doi.org/10.1101/sqb.1986.051.01.032
- Slamon DJ, Leyland-Jones B, Shak S, Fuchs H, Paton V, Bajamonde A, et al. Use of Chemotherapy plus a Monoclonal Antibody against HER2 for Metastatic Breast Cancer That Overexpresses HER2. N Engl J Med. [Internet]. 2001;344(11):783-92. Disponible en: https://doi.org/10.1056/nejm200103153441101
- Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. [Internet]. 1977;74(12):5463-7. Disponible en: https://doi.org/10.1073/pnas.74.12.5463
- International Human Genome Sequencing Consortium, Whitehead Institute for Biomedical Research, Center for Genome Research:, Lander ES, Linton LM, Birren B, Nusbaum C, et al. Initial sequencing and analysis of the human genome. Nature. [Internet]. 2001;409(6822):860-921. Disponible en: https://doi.org/10.1038/35079657
- Vogelstein B, Kinzler KW. The multistep nature of cancer. Trends in Genetics. [Internet]. 1993;9(4):138-41. Disponible en: https://doi.org/10.1016/0168-9525(93)90209-z
- Rubin MA, Demichelis F. The Genomics of Prostate Cancer: A Historic Perspective. Cold Spring Harb Perspect Med. [Internet]. 2019;9(3):a034942. Disponible en: https://doi.org/10.1101/cshperspect.a034942
- Casolino R, Beer PA, Chakravarty D, Davis MB, Malapelle U, Mazzarella L, et al. Interpreting and integrating genomic tests results in clinical cancer care: Overview and practical guidance. CA Cancer J Clin. [Internet]. 2024;74(3):264-85. Disponible en: https://doi.org/10.3322/caac.21825
- Brlek P, Bulić L, Bračić M, Projić P, Škaro V, Shah N, et al. Implementing Whole Genome Sequencing (WGS) in Clinical Practice: Advantages, Challenges, and Future Perspectives. Cells. [Internet]. 2024;13(6):504. Disponible en: https://doi.org/10.3390/cells13060504
- Heather JM, Chain B. The sequence of sequencers: The history of sequencing DNA. Genomics. [Internet]. 2016;107(1):1-8. Disponible en: https://doi.org/10.1016/j.ygeno.2015.11.003
- Torres-Narvaez ES, Mendivelso-González DF, Artunduaga-Alvarado JA, Ortega-Recalde O. Cancer genomics and bioinformatics in Latin American countries: applications, challenges, and perspectives. Front Oncol. [Internet]. 2025;15:1584178. Disponible en: https://doi.org/10.3389/fonc.2025.1584178
- Wang D, Liu B, Zhang Z. Accelerating the understanding of cancer biology through the lens of genomics. Cell. [Internet]. 2023;186(8):1755-71. Disponible en: https://doi.org/10.1016/j.cell.2023.02.015
- Berger MF, Mardis ER. The emerging clinical relevance of genomics in cancer medicine. Nat Rev Clin Oncol. [Internet]. 2018;15(6):353-65. Disponible en: https://doi.org/10.1038/s41571-018-0002-6
- De Las Salas B, Sánchez N, Gutiérrez L, Pérez S, Mercado V, Becerra P, et al. Investigación clínica en la era de la inmunoterapia y la genómica. Med. [Internet]. 2024;46(2):372-92. Disponible en: https://doi.org/10.56050/01205498.2371
- Sakamoto Y, Sereewattanawoot S, Suzuki A. A new era of long-read sequencing for cancer genomics. J Hum Genet. [Internet]. 2020;65(1):3-10. Disponible en: https://doi.org/10.1038/s10038-019-0658-5
- Valenti F, Falcone I, Ungania S, Desiderio F, Giacomini P, Bazzichetto C, et al. Precision Medicine and Melanoma: Multi-Omics Approaches to Monitoring the Immunotherapy Response. IJMS. [Internet]. 2021;22(8):3837. Disponible en: https://doi.org/10.3390/ijms22083837
- Testa U, Castelli G, Pelosi E. Breast Cancer: A Molecularly Heterogenous Disease Needing Subtype-Specific Treatments. Med Sci (Basel). [Internet]. 2020;8(1):18. Disponible en: https://doi.org/10.3390/medsci8010018
- Liskova A, Samec M, Koklesova L, Giordano FA, Kubatka P, Golubnitschaja O. Liquid Biopsy is Instrumental for 3PM Dimensional Solutions in Cancer Management. JCM. [Internet]. 2020;9(9):2749. Disponible en: https://doi.org/10.3390/jcm9092749
- Moldogazieva NT, Zavadskiy SP, Terentiev AA. Genomic Landscape of Liquid Biopsy for Hepatocellular Carcinoma Personalized Medicine. Cancer Genomics Proteomics. [Internet]. 2021;18(3 Suppl):369-83. Disponible en: https://doi.org/10.21873/cgp.20266
- Cammarata G, De Miguel-Perez D, Russo A, Peleg A, Dolo V, Rolfo C, et al. Emerging noncoding RNAs contained in extracellular vesicles: rising stars as biomarkers in lung cancer liquid biopsy. Ther Adv Med Oncol. [Internet]. 2022;14:17588359221131229. Disponible en: https://doi.org/10.1177/17588359221131229
- Black JRM, McGranahan N. Genetic and non-genetic clonal diversity in cancer evolution. Nat Rev Cancer. [Internet]. 2021;21(6):379-92. Disponible en: https://doi.org/10.1038/s41568-021-00336-2
- Mustachio LM, Roszik J. Single-Cell Sequencing: Current Applications in Precision Onco-Genomics and Cancer Therapeutics. Cancers. [Internet]. 2022;14(3):657. Disponible en: https://doi.org/10.3390/cancers14030657
- Bowes AL, Tarabichi M, Pillay N, Van Loo P. Leveraging single‐cell sequencing to unravel intratumour heterogeneity and tumour evolution in human cancers. The Journal of Pathology. [Internet]. 2022;257(4):466-78. Disponible en: https://doi.org/10.1002/path.5914
- Mulder NJ, Adebiyi E, Adebiyi M, Adeyemi S, Ahmed A, Ahmed R, et al. Development of Bioinformatics Infrastructure for Genomics Research. Glob Heart. [Internet]. 2017;12(2):91-98. Disponible en: https://globalheartjournal.com/article/10.1016/j.gheart.2017.01.005
- The ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Nature. [Internet]. 2020;578(7793):82-93. Disponible en: https://doi.org/10.1038/s41586-020-1969-6
- Pan D, Jia D. Application of Single-Cell Multi-Omics in Dissecting Cancer Cell Plasticity and Tumor Heterogeneity. Front Mol Biosci. [Internet]. 2021;8:757024. Disponible en: https://doi.org/10.3389/fmolb.2021.757024
- Neinavaie F, Ibrahim-Hashim A, Kramer AM, Brown JS, Richards CL. The Genomic Processes of Biological Invasions: From Invasive Species to Cancer Metastases and Back Again. Front Ecol Evol. [Internet]. 2021;9:681100. Disponible en: https://doi.org/10.3389/fevo.2021.681100
- Williams ST, Wells G, Conroy S, Gagg H, Allen R, Rominiyi O, et al. Precision oncology using ex vivo technology: a step towards individualised cancer care? Expert Rev Mol Med. [Internet]. 2022;24:e39. Disponible en: https://doi.org/10.1017/erm.2022.32
- Cancer Genomics: The Road Ahead. Cell. [Internet]. 2013;155(1):9-10. Disponible en: https://linkinghub.elsevier.com/retrieve/pii/S0092867413011513
- McVeigh TP, Hughes LM, Miller N, Sheehan M, Keane M, Sweeney KJ, et al. The impact of Oncotype DX testing on breast cancer management and chemotherapy prescribing patterns in a tertiary referral centre. European Journal of Cancer. [Internet]. 2014;50(16):2763-70. Disponible en: https://doi.org/10.1016/j.ejca.2014.08.002
- Allemand C, Valerio AC, Calvo MF, Izbizky G, McLean I, Terrier F, et al. Impacto del Score de Recurrencia de 21 genes (Oncotype DX®) sobre la toma de decisión en tratamiento adyuvante: un estudio multicéntrico y colaborativo / Impact of the 21-gene Recurrence Score (Oncotype DX®) on decision-making in adjuvant treatment: a multicenter and collaborative study. Rev Argent Mastología [Internet]. 2024;42(154):13-27. Disponible en: https://doi.org/10.29289/2594539420210026
- Sparano JA, Gray RJ, Makower DF, Pritchard KI, Albain KS, Hayes DF, et al. Adjuvant Chemotherapy Guided by a 21-Gene Expression Assay in Breast Cancer. N Engl J Med. [Internet]. 2018;379(2):111-21. Disponible en: https://doi.org/10.1056/NEJMoa1804710
- Carugo A, Draetta GF. Academic Discovery of Anticancer Drugs: Historic and Future Perspectives. Annu Rev Cancer Biol. [Internet]. 2019;3(1):385-408. Disponible en: https://doi.org/10.1146/annurev-cancerbio-030518-055645
- Pich O, Bailey C, Watkins TBK, Zaccaria S, Jamal-Hanjani M, Swanton C. The translational challenges of precision oncology. Cancer Cell. [Internet]. 2022;40(5):458-78. Disponible en: https://doi.org/10.1016/j.ccell.2022.04.002
- O’Loughlin TA, Gilbert LA. Functional Genomics for Cancer Research: Applications In Vivo and In Vitro. Annu Rev Cancer Biol. [Internet]. 2019;3(1):345-63. Disponible en: https://doi.org/10.1146/annurev-cancerbio-030518-055742
- Kabadi A, McDonnell E, Frank CL, Drowley L. Applications of Functional Genomics for Drug Discovery. SLAS Discov. [Internet]. 2020;25(8):823-42. Disponible en: https://doi.org/10.1177/2472555220902092
- Li H, Yang Y, Hong W, Huang M, Wu M, Zhao X. Applications of genome editing technology in the targeted therapy of human diseases: mechanisms, advances and prospects. Sig Transduct Target Ther. [Internet]. 2020;5(1):1. Disponible en: https://doi.org/10.1038/s41392-019-0089-y
- Afolabi LO, Afolabi MO, Sani MM, Okunowo WO, Yan D, Chen L, et al. Exploiting the CRISPR‐Cas9 gene‐editing system for human cancers and immunotherapy. Clin & Trans Imm. [Internet]. 2021;10(6):e1286. Disponible en: https://doi.org/10.1002/cti2.1286
- Tran B, Dancey JE, Kamel-Reid S, McPherson JD, Bedard PL, Brown AMK, et al. Cancer Genomics: Technology, Discovery, and Translation. JCO. [Internet]. 2012;30(6):647-60. Disponible en: https://doi.org/10.1200/jco.2011.39.2316
- Vera J, Lai X, Baur A, Erdmann M, Gupta S, Guttà C, et al. Melanoma 2.0. Skin cancer as a paradigm for emerging diagnostic technologies, computational modelling and artificial intelligence. Briefings in Bioinformatics. [Internet]. 2022;23(6):bbac433. Disponible en: https://doi.org/10.1093/bib/bbac433
- Valent P, Orfao A, Kubicek S, Staber P, Haferlach T, Deininger M, et al. Precision Medicine in Hematology 2021: Definitions, Tools, Perspectives, and Open Questions. HemaSphere. [Internet]. 2021;5(3):e536. Disponible en: https://doi.org/10.1097/hs9.0000000000000536
- Ruíz-Patiño A. Futuro de la oncología personalizada y el diagnóstico molecular. Med. [Internet]. 2024;46(2):442-50. Disponible en: https://doi.org/10.56050/01205498.2375